预测新冠病毒“蛋白质折叠”重磅武器:AlphaFold!精度碾压生物与病毒专家
欢迎关注“创事记”微信订阅号:sinachuangshiji
作者/新智元
来源:新智元(ID:AI_era)
谷歌母公司Alphabet旗下的明星AI公司DeepMind以打败人类享誉世界,凭借AlphaGo击败人类围棋世界冠军,凭借AlphaStar击败99.8%的人类玩家。而这一次,DeepMind要帮助人类击败新冠病毒。

要想检测病毒并开发疫苗,科学家必须首先了解病毒,特别是病毒蛋白质的结构。这是一个漫长的过程,需要几个月的时间,并且有时候是徒劳的。近年来,研究人员已经转向计算机预测。
世界各地的实验室正在研究冠状病毒,DeepMind的深度学习系统叫做“AlphaFold”。
日前,DeepMind发布了一篇文章,表示希望通过“发布与SARS-CoV-2(引起COVID-19的病毒)相关的几种未被充分研究的蛋白质的结构预测”来帮助进行这项研究。接下来先来和大家分享DeepMind的这篇文章。
DeepMind:COVID-19相关蛋白结构的计算预测
“人类对冠状病毒的研究已经有几十年的历史,所以利用以往的数据库对COVID-19疫情能做出较快的反应,短短几天就研究出了新病毒的检测方法。
而此次COVID-19的罪魁祸首SARS-CoV-2病毒相关的蛋白质结构一直未能确定,传统的方法可能需要几个月甚至更长时间,这对我们理解病毒的功能和传播机制造成了很大的障碍。
鉴于传统方法耗时过长,我们将新版的AlphaFold用于SARS-CoV-2相关的蛋白质结构预测,新系统可以在没有类似蛋白质结构的前提下,得出精确预测。
我们分享了几种模型预测的病毒蛋白质结构,希望能为广大研究人员提供一些帮助。
我们相信新系统比我们早期的CASP13系统更准确。 此前,我们成功预测了一个蛋白质数据库中经过实验验证的SARS-CoV-2棘突蛋白结构,这给了我们足够的信心,新系统预测其他蛋白质结构也是有可能的。

最近,我们与英国弗朗西斯·克里克研究院的几位同事分享了我们的研究结果,其中包括结构生物学家和病毒学家,他们鼓励我们现在就公布我们的结构。 我们的模型会指出结构的哪些部分更有可能是正确的。 虽然这些未被研究的蛋白质不是当前治疗的重点,但它们可能会增加研究人员对SARS-CoV-2的理解。
正常情况下,我们会等到这项工作经过同行评审再发表。 但是鉴于时间的紧迫性和疫情的严重程度,我们决定发布SARS-CoV-2相关的六种蛋白质的预测结构,这些数据文件采用开源的许可证,现在任何人都可以使用了。
有兴趣的研究人员可以通过我们提供的链接下载这些数据文件,其中还附带了很多技术细节。 最后还要强调一下,这些都是预测的结构,还没有经过实验验证。”
新智元根据DeepMind文章中提供的下载文件制作出了六种蛋白质的预测结构图,仅供参考(不保证100%精确):

原文件下载地址:
https://storage.googleapis.com/deepmind-com-v3-datasets/alphafold-covid19/structures_4_3_2020.zip
DeepMind预测新冠病毒“蛋白质折叠”的重磅武器:AlphaFold!精度碾压友商和人类
预测出新冠病毒的蛋白质结构至关重要,可以使科学家能够通过模拟和模型获得更多关于蛋白质形状及其运作方式的知识,也给新药物的开发开辟了新的潜力,降低了实验成本,并极大的加快科学家找出更有效治疗方法的流程,最终得以拯救全世界患者。
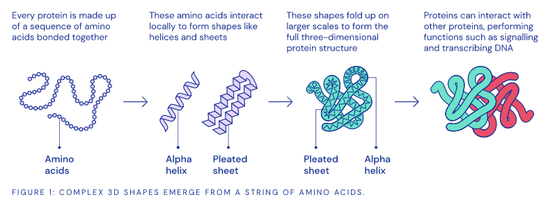
而此次DeepMind用来预测新冠病毒蛋白质结构的重磅武器,就是其在2018年底推出的、被各路媒体热捧的AlphaFold。

DeepMind汇集了来自结构生物学、物理学和机器学习领域的专家,以应用尖端技术,仅根据其基因序列预测蛋白质的3D结构。
据悉,在有蛋白质结构预测的“奥运会”之称CASP全球竞赛上,AlphaFold不仅在友商中遥遥领先,甚至其预测的精度已经碾压了人类专家。
纯粹从基因序列中确定蛋白质的三维形状是一项复杂的任务,挑战在于DNA仅包含有关蛋白质结构块序列的信息,这些结构块称为氨基酸残基,排布形成长链。预测这些长链按照何种方式折叠成蛋白质的复杂3D结构,就是所谓的“蛋白质折叠问题”。
蛋白质越大,模型就越复杂和困难,因为氨基酸之间的相互作用更加复杂。有研究人员估计,以现有的条件的水平,要想准确确定全部蛋白质的3D结构,需要的时间可能比宇宙的寿命还要长。
基于深度学习的蛋白质结构预测新方法
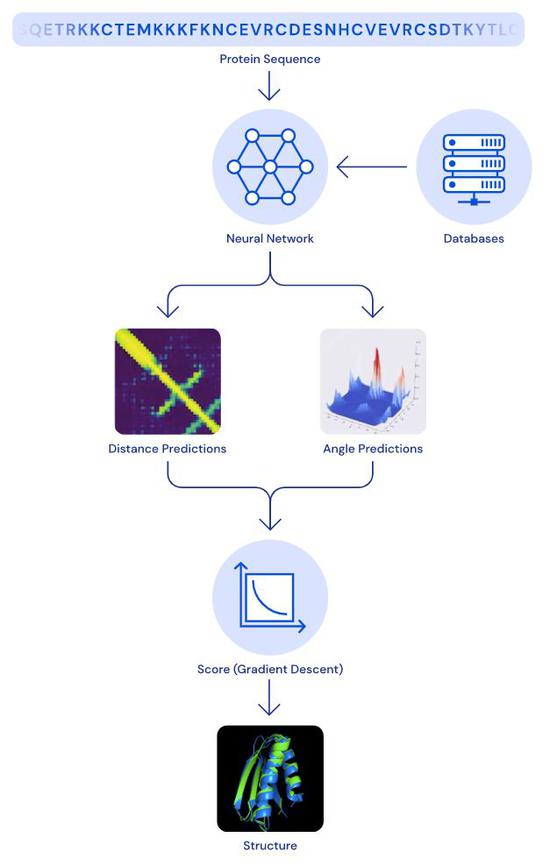
AlphaFold从头开始对蛋白质的形态结构进行建模,而没有使用已经解析的蛋白质作为模板。结果在预测蛋白质结构的物理性质时达到高准确度,在此基础上使用两种不同的方法,来构建完整蛋白质结构的预测。
这两种方法都依赖于深度神经网络,可以从蛋白质的基因序列中预测其特性。该网络预测指标主要有两个:(1)氨基酸对之间的距离(2)连接这些氨基酸的化学键之间的角度。该技术用于估计氨基酸对之间是否彼此接近。
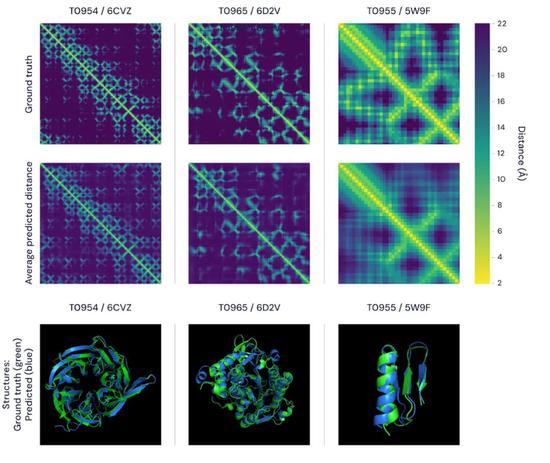
上图具有三种蛋白质的距离矩阵。每个像素的亮度代表着蛋白质序列中氨基酸之间的距离。像素越亮,配对越近。在顶部行中显示的是真实的,实验确定的距离,而在底部行中显示的是Alphafold的预期距离分布的平均值。重要的是,这些匹配在全球范围和地方范围都很好。底部面板使用3D模型来表示相同的比较,其特征是阿尔法福尔德的预测(蓝色)与地面真实数据(绿色)相对于相同的三种蛋白质。
DeepMind团队训练神经网络,预测蛋白质中每对残基之间的距离的单独分布。然后将这些概率进行组合,形成相应蛋白质结构预测的准确度分数。此外还训练了一个单独的神经网络,将所有的预测距离汇总,估计出预测结构与实际结构间的接近程度。
使用这些评分函数,可以找到与预测相匹配的蛋白质结构。第一种方法建立在结构生物学中常用的技术基础上,用新的蛋白质结构片段反复替换原蛋白质结构片段。为此,研究团队训练了一个生成神经网络来发明新的蛋白质片段,不断提高预测的蛋白质结构的得分。
第二种方法通过梯度下降来优化得分。梯度下降是机器学习中常用的技术,可用于实现微小的,渐进式的改进,最终产生高精确的结构。研究人员将该技术用于整个蛋白质长链上,而不是在组装之前必须单独折叠的碎片上,降低了预测过程的复杂度。
等不及同行评审,第一时间发布预测结果,准确度未知
和之前GPT-2捂着不发布形成鲜明对比,由于当前疫情的急迫性,DeepMind等不及通过同行评审,第一时间就发布了蛋白质结构的预测结果,并以pdb文件存放,研究者下载后可自行利用pdb文件中的数据绘制图像。
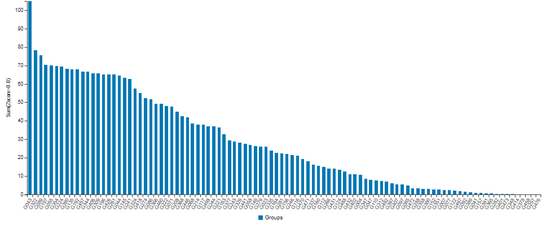
目前看来,AlphaFold虽然在比赛中获得了优异的成绩,但其在预测的稳定性方面的缺陷也是非常显著的。在比赛中表现出两个极端:43项预测中,25个预测的非常准确,而其他则错的离谱。因此AlphaFold对新冠病毒的预测结果的准确度仍是未知数,期待同行评审结果,以及实际临床治疗的验证。
不管怎样,DeepMind此举为AI应用到实际场景,尤其是应用于抗疫斗争中,开辟了一条新的思路。同时也期待更多的国内的AI企业能够在此次抗疫斗争中,找到自己的突破口!

(声明:本文仅代表作者观点,不代表新浪网立场。)