万物皆硬盘!一只“兔子”实证DNA存储无所不在

欢迎关注“创事记”微信订阅号:sinachuangshiji
原标题:万物皆硬盘!一只“兔子”实证DNA存储无所不在,中国已列为重点专项|专家解读
文/孙滔
来源:DeepTech深科技(ID:deeptechchina)
DNA硬盘将颠覆人们对数据存储的认知。
在我们目前的存储世界里,硬盘必须是硬盘的样子,磁带必须是磁带的形状,光盘也须是光盘的外形,而DNA硬盘则不受形状所限。
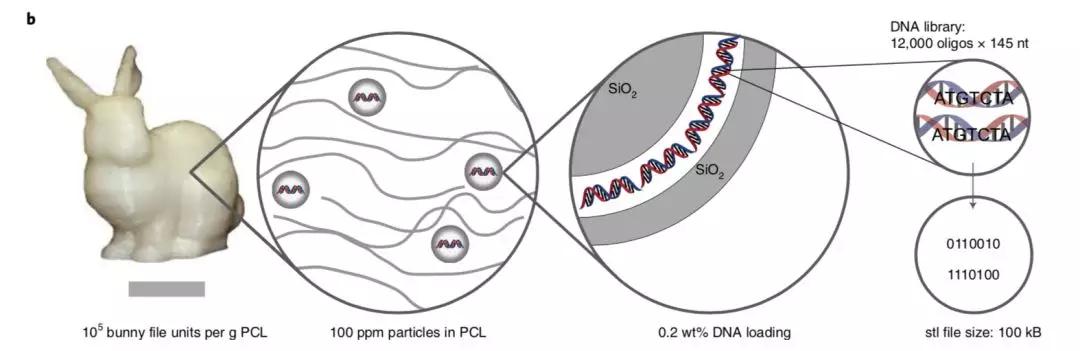
为了证实这个说法,科学家用3D打印制作了一只兔子,并且这只兔子三维结构的数据以双链DNA结构的形式内置在打印材料中。也就是说,通过编码和解码,这只兔子模型实现了其自身数据的DNA存储和传递。推而广之,世界万物皆可实现DNA存储。
简单说,数据写入即是人工合成DNA,数据读取即是DNA测序,数据的拷贝即是DNA的复制。
 动图|DNA数据存储拓展了将信息直接嵌入日常物品的可能性。(来源:《连线》)
动图|DNA数据存储拓展了将信息直接嵌入日常物品的可能性。(来源:《连线》)今天(北京时间12月10日),这项研究发表在了《自然-生物技术》(NatureBiotechnology)期刊上,通讯作者系MyHeritage首席科学家、哥伦比亚大学副教授YanivErlich和苏黎世联邦理工学院功能材料实验室教授RobertGrass。
Yaniv Erlich 将作为演讲嘉宾(点击了解详情)出席12月13日-14日在北京举办的第三届EmTechChina全球新兴科技峰会,与我们分享MyHeritage在生命科学领域取得的最新进展。他表示,会在北京演讲现场向观众展示这只“兔子”。
首只DNA存储数据的兔子
 图|包含了DNA数据的斯坦福兔子。(来源:苏黎世联邦理工学院)
图|包含了DNA数据的斯坦福兔子。(来源:苏黎世联邦理工学院)这里需要提到一个概念,斯坦福兔子(StanfordBunny)。这不是某个艺术工作者随意的作品,而是一种计算机图形学领域广泛采用的3D测试模型,在1994年于斯坦福大学制作。
研究人员将斯坦福兔子的0和1的二进制数据转换为DNA中4种碱基的数据(A、T、C、G),进而将DNA片段封装在二氧化硅小球内(小球大小为160纳米),这些小球则被嵌入可生物降解的热塑性聚酯中,最后使用所得的热塑性聚酯来进行兔子的3D打印。
这是一个DNA存储编码的过程,由压缩、纠错和转换3部分组成。在转换为DNA数据之前,斯坦福兔子的二进制立体光刻文件大小为100KB,用以合成DNA编码的数字蓝图被压缩到了45KB,这是为了最大化地利用DNA存储空间,需要对信息去除冗余以达到压缩的目的。
每个寡核苷酸长度为145个核苷酸,由104个有效片段与41个核苷酸的聚合酶链式反应(PCR)退火位点组成。随后研究人员利用DNA喷泉编码技术(DNAFountain),将数码信息转换为DNA序列信息,即1.2万个DNA寡核苷酸,再将PCR扩增的寡核苷酸封装到二氧化硅小球内,每个小球包含了数十个合成DNA分子。当然这个组装采用体外人工合成的方式,这样可避免细胞的排外性及受生物活动的影响。将DNA链封装于二氧化硅小球中也是为了防止DNA降解。

图|斯坦福兔子的3D打印与解码原理图,分为二进制数据转换为DNA数据、DNA封装、嵌入热塑性聚酯、打印、DNA解码等过程。(来源:《自然-生物技术》)
 图|斯坦福兔子的还原结构。(来源:《自然-生物技术》)
图|斯坦福兔子的还原结构。(来源:《自然-生物技术》)那么,如何解码呢?其原理是利用PCR技术对存储的DNA片段进行复制扩增以备份,再对扩增得到的DNA片段进行测序,获取碱基序列后对序列纠错、去冗余、解码,即可得到原始信息。
在这个研究中,研究人员利用存储在兔子中的DNA来复制兔子数据。具体而言,研究人员从兔子耳朵处剪下10毫克的打印材料,这占兔子总重量3.2克的0.3%,然后提取出其中的DNA(至此需4小时),扩增并测序(需17小时)。尽管有5.9%的原始寡核苷酸丢失以及存在测序误差,但研究人员采用DNA喷泉解码器完美解读了斯坦福兔子的数据。解码过程只需要在普通笔记本电脑运行数分钟即可完成。
那么由此循环,由前一代扩增的DNA被封装到下一代中,研究人员连续创造出了5代兔子,且没有任何信息损失。即使第四代和第五代之间相隔了9个月,DNA信息一直保持高保真性和稳定性。
为了扩展这个研究的场景,研究人员还将一段有关华沙犹太区档案的视频编码进树脂玻璃中,再用该树脂玻璃制造了一副眼镜。而只需一小块树脂玻璃,就能恢复其中隐藏的信息。
YanivErlich对DeepTech表示,这项研究最大的突破在于实证了万物皆可实现DNA存储的理论,且不受任何形状限制。
1克DNA能存储2.2亿部电影
DNA数据存储的密度之高令人难以置信。有数据称,1克DNA即可储存215PB的信息,而硬盘的存储量不过几个T。要知道,1PB=1024TB,而1TB=1024GB,按照高清电影每部10GB算,1克DNA能够存储2.2亿部电影。
DNA信息的读取不涉及兼容问题,且DNA是可降解的材料,相比其他存储介质更加环保。此外DNA对于高温、震荡等外部环境具有极强的抗干扰能力。
鉴于以上特点,加上DNA是按顺序编码存储信息的,且存储信息段存在起始点和终止点,还可以引入纠错码确保信息的完整性,于是,DNA就成了数据存储研究领域的宠儿。尤其是那些不常用但却需要长期保存的冷数据,例如政府文件、历史档案等尤其适合DNA存储方式。
自从1950年代DNA双螺旋结构被发现以来,科学家就萌生了用DNA的4种碱基来存储数据的想法。哈佛大学的GeorgeChurch教授团队于2012年将一本 Church著作的图书数据(659kB)存储在了DNA中,他们采用了二对一的对应关系,其中二进制的“0”用腺嘌呤或胞嘧啶表示,而二进制的“1”则用鸟嘌呤或胸腺嘧啶代表。
2017年,YanivErlich等人在《科学》杂志上报告说,他们将6个文件存入了DNA中,这6个文件包括一个完整的计算机操作系统、一种计算机病毒、一部法国电影,和由信息论创始人、美国数学家香农(ClaudeShannon)在1948年进行的一项研究。
在《科学》杂志的这项研究中,YanivErlich正是采用了斯坦福兔子研究中用到的DNA喷泉编码技术,即将DNA片段随机打包为“水滴”来储存,这些水滴中添加了额外的标签以便以后能够重新组装。该技术具有独立随机性,且编译码复杂程度低,有容错纠错机制,能高概率恢复存储信息。
“万物DNA”的障碍:成本高、时间效率低
研究人员将这个兔子硬盘的开发方式称为“万物DNA”(DNA-of-things,DoT)存储架构,它可以生成具有不变记忆的材料。
那么这个“万物DNA”存储架构有什么应用呢?YanivErlich等人在《自然-生物技术》论文中举例称,在3D医学或牙科植入物领域,因为每个结构都是唯一的,那么就可以根据患者的精确解剖结构进行定制。鉴于二氧化硅小球是无毒的,那么就可以将植入物的设计信息和其他医学信息都存储其中,如此就产生了长期的电子病历备份,而传统的电子病历通常只保留5年到10年。
此外,作者认为此技术还可用于建筑、药品和电子元件等冷数据的存储。
“万物DNA”的另外一个应用是信息密写术。因为各种日常物品均可以作为秘密数据的携带者,那么数据盗窃者就面临多重破解障碍:首先,因为二氧化硅小球不会改变储存介质的特性,那么破解者须测试多个物品才可能找到存储介质。其次,因为DNA被隔离在二氧化硅小球中,那么普通的DNA传感技术如紫外线将不能检测出DNA。再次,即使破解者恢复了DNA文库,也需要找到退火位点才能通过PCR来扩增信息。
作者还认为,该技术强大的自我复制能力大有可为,它为本地化数据存储和离线存储找到了好思路。
目前,相关技术已有专利布局。YanivErlich持有DNA存储领域的专利;苏黎世联邦理工学院拥有DNA封装的专利;YanivErlich和RobertGrass是“万物DNA”(DoT)专利申请的发明人。
这只兔子成本高昂,完成DNA数据存储大约需14000元。对于成本问题,作者认为,虽然对于定制物品,DNA合成的成本仍然较高,但若要量产,DNA文库的合成成本将变得微不足道。
不过,成本仍是障碍。尽管DNA合成和测序的成本每年均在呈指数下降,有数据称从2002年的218750元/兆降至了2016年的4.41元/兆,但相对于普通硬盘存储而言,这仍然费用高昂。
实际上,DNA存储距离消费级应用尚远的更重要原因是时间效率低下。YanivErlich说,“如果要应用到普通消费者,还需要实现测序仪的便携化,以及样品的极高效制备。”
从斯坦福兔子的编码和解码过程可知,整个过程需要数十个小时。这就意味着,若要扩大DNA的存储应用范围,除了降低成本还需实现像硬盘、磁带那样随时随地写入或读取信息的性能。
北京大学信息学院副研究员张成长期从事DNA分子计算和纳米智能领域的研究,包括DNA计算与存储、分子电路、自组装纳米孔器件和纳米智能机器人等方面的研究。他告诉DeepTech,DNA存储发展道路上最大的障碍是输入和读取的效率,时间成本是一个非常大的问题。“我可以在实验室里花上一周时间来解码,但没有哪个普通消费者愿意这样等,而只要涉及到DNA扩增,这个时间要求就是必然的。”
这就需要DNA编码、存储和解码均实现便携化。随着便携式DNA测序仪的进一步发展,可能会实现随时DNA测序。然而这只是解码的环节。
中国重点专项已在布局
目前,DNA存储在国内属于新兴领域,革命性突破还需要领域内科学家共同努力。张成说,DNA存储这个领域实际上是在2016年以后才开始加快步伐的,至于国内发展不够快的情况有两方面原因。其一是该研究领域的门槛非常高,需要计算机科学、生物学、化学等多领域协作。其二是DNA存储技术虽然有广泛的应用前景,但目前仍然存在时间上和成本上巨大的挑战,“何时能真正意义上走入商业市场,还有赖于相关前沿DNA纳米技术的发展”。
已经有大公司盯上了DNA存储。自2015年起,微软研究院与华盛顿大学的研究人员合作就开始开展DNA数据存储研究,希望将合成DNA变成耐用、易操作的高密度信息存储介质。
2016年,研究团队成功地将4个图像文件信息存储到一段人造DNA片段上,并完好无损地将它们取了出来。
2019年3月,他们首次实现了全自动的DNA数据存储与提取。在这项实验中,研究团队开发了全自动的端到端系统,在合成DNA片段中写入“hello”一词,并将DNA上的数据转换回了通用的数字信息。微软称,这项自动化技术是让DNA数据存储得以走出实验室,应用到商业数据中心的一个重要的里程碑。
实际上,中国政府也在这个领域加大支持。根据科技部、深圳市人民政府《部市联动组织实施国家重点研发计划“合成生物学”重点专项框架协议》,中央财政和深圳市联合出资,共同组织实施“合成生物学重点专项”。
在《“合成生物学”重点专项2018年度项目申报指南》中对项目“使用合成DNA进行数据存储的技术研发”是这么描述的:
研究内容:开发利用合成DNA高效快速、高密度数据加密编码转码,随机读取,无损解读新方法;开发多类型数据存储DNA介质;通过合成DNA开发快速编码,存储及数据读取的集成型软件系统。
考核指标:开发1套DNA数据编码算法,实现数据信息到DNA码的高密度存储(单位编码效率bits/base>1.6);开发1套DNA纠错及索引算法,实现数据无损解读;开发1套分区及随机读取流程,实现DNA数据存储的随存随取;开发1套适用不同类型数据到DNA序列转换算法。
据今年7月来自南方科技大学的信息,科技部公示了国家重点研发计划“合成生物学”重点专项2018年度拟立项项目清单,南方科技大学生物医学工程系系主任蒋兴宇教授作为项目负责人的项目“使用合成DNA进行数据存储的技术研发”成功入选,其牵头的“使用合成DNA进行数据存储的技术研发”项目总经费达2203万元。
该项目由南方科技大学牵头,上海交通大学、中国科学院长春应用化学研究所、福州大学、同济大学联合申报。项目拟通过发展新型存储技术以应对大数据的爆炸式增长,解决数据快速增长与数据有效存储和利用之间的矛盾,推动中国在DNA数据存储基础研究领域的原始创新和科学突破。
“由于DNA存储技术领域的强学科交叉性,必须依靠计算机、生物、化学、数学和其他多个相关学科的协同发展,才有可能使我国在DNA存储的国际竞争中占据先机“,张成指出。
面对DNA存储这种颠覆性技术的历史机遇,多学科交叉和国际化协作必不可少。比如,该文的通讯作者YanivErlich,其2017年在哥伦比亚大学的计算机学院发表 Science 文章,设计DNA喷泉算法;而另一通讯作者是苏黎世联邦理工学院功能材料实验室的Titulary教授RobertGrass,他负责将DNA封装到玻璃材料中。
张成说,“作为国内科研工作者,我们也同样非常注重学科交叉。”北京大学许进-张成联合课题组的计算机专业学生,不仅熟悉编程等计算机技术,同时还能够走入生化实验室,进行精密的DNA纳米技术实验操作。例如,通过学科交叉,2019年联合课题组构建的可循环DNA电路的工作就发表在《美国化学会志》(JACS)上,这是北京大学软件所历史上的首次跨学科 JACS 论文。
张成坦言,目前DNA存储相关研究仍在实验室探索阶段,属于基础研究阶段。因此,国家的相关政策引导和支持,对于DNA存储在中国的发展至关重要。